Maschinenlesbare chemische Strukturen
Einleitung
Die Suche nach relevanten Artikeln auf der Grundlage von IUPAC-Namen oder Trivialnamen von Molekülen kann eine schwierige und zeitraubende Aufgabe sein, während chemische Strukturidentifikatoren eine eindeutige Identifizierung von Verbindungen ermöglichen. Das Neuzeichnen chemischer Strukturen ist arbeits- und zeitintensiv, während CTfiles wie Molfiles ohne zusätzlichen Aufwand mit jeder gängigen Strukturzeichnungssoftware verwendet werden können.
Die Bereitstellung maschinenlesbarer chemischer Strukturen als CT-Dateien wie Molfiles, InChI-Strukturidentifikatoren und SMILES-Strukturcodes als Teil eines mit einem Forschungsartikel verbundenen Datensatzes verbessert dessen Auffindbarkeit, indem der Artikel leicht indizierbar und nach Strukturen durchsuchbar wird. Dies verbessert auch die Interoperabilität und erleichtert die Wiederverwendung der wissenschaftlichen Ergebnisse.
Während solche Informationen für strukturierte fachspezifische Repositorien wie Chemotion Repository nicht erforderlich sind, da diese Informationen von der Repository-Software bzw. Chemotion ELN generiert werden, profitieren Datensätze in generischen Repositorien von diesen Informationen.
Im Folgenden wird eine Anleitung gegeben, wie die Maschinenlesbarkeit von Chemie-Forschungsartikeln erhöht werden kann, indem maschinenlesbare chemische Strukturen innerhalb des zugehörigen Datensatzes bereitgestellt werden. Darüber hinaus werden Empfehlungen zur Bereitstellung von Strukturcodes und Identifikatoren in einer maschinenlesbaren Zusatztabelle innerhalb des zugehörigen Datensatzes, publiziert in einem generischen Repositorium, gegeben.
Mol-Dateien abrufen
Alle gängigen Strukturzeichenprogramme speichern Mol-Dateien. Kopieren Sie die Struktur in ein neues Dokument in Ihrer bevorzugten Strukturzeichensoftware. Wählen Sie dann Datei -> Speichern unter -> wählen Sie MDL Molfile aus dem Dropdown-Menü -> Speichern.
Der Name der Datei kann in Anlehnung an Ihren Laborjournaleintrag und zusätzlich die Nummerierung der Struktur im Artikel gewählt werden.
SMILES, InChI und InChIKey abrufen
ChemDoodle
Um SMILES, InChI und InChIKey in ChemDoodle abzurufen, markieren Sie eine Struktur und wählen dann Edit -> Copy As -> Daylight SMILES oder IUPAC InChI.
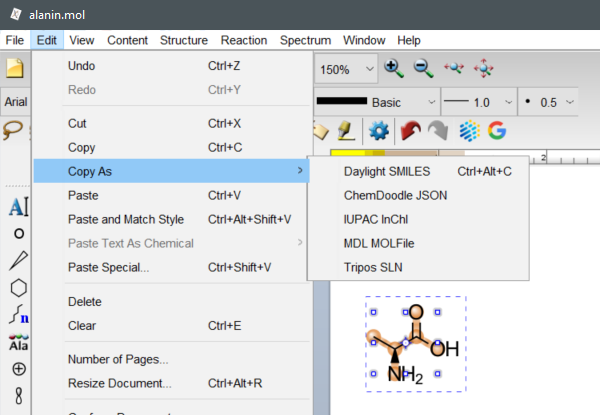
(ChemDoodle v11.7.0, iChemLabs, LCC., Chesterfield, VA, Vereinigte Staaten, 2021.)
Sie können auch eine Struktur auswählen und dann Struktur -> Generate Line Notation -> Daylight SMILES oder IUPAC InChI wählen.
Alternativ können Sie SMILES, InChI und InChIKey auch als Textdateien speichern, indem Sie File -> Save as -> Daylight SMILES oder InChI aus dem Dropdown-Menü wählen und anschließend -> Speichern.
Wenn Sie "IUPAC InChI" wählen, wird auch der InChIKey angezeigt, wenn er in den Voreinstellungen aktiviert ist. Um InChIKey einzuschließen, wählen Sie Edit -> Preferences -> Files tab -> scrollen Sie nach unten und markieren Sie InChI key einschließen.
ChemDraw Professional
Um SMILES, InChI und InChIKey in ChemDraw Professional abzurufen, markieren Sie eine Struktur und wählen dann Edit -> Copy As -> SMILES, InChI oder InChIKey.
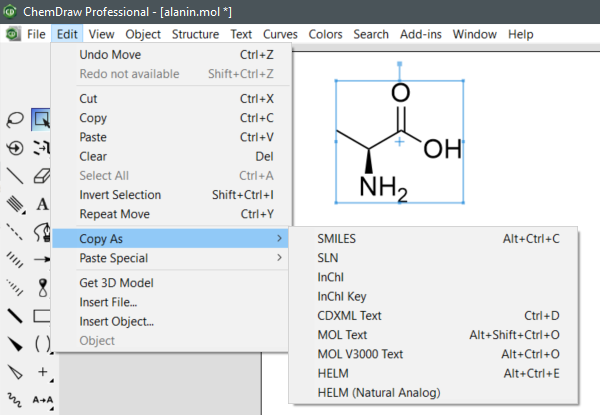 "
"
(ChemDraw Professional v20.1.0.11, PerkinElmer Informatics, Inc., Waltham, MA, Vereinigte Staaten, 2021.)
ChemSketch
Um SMILES, InChI und InChIKey in ACD/ChemSketch abzurufen, markieren Sie eine Struktur und wählen Sie dann Tools -> Generate -> SMILES Notation oder InChI for Structure.
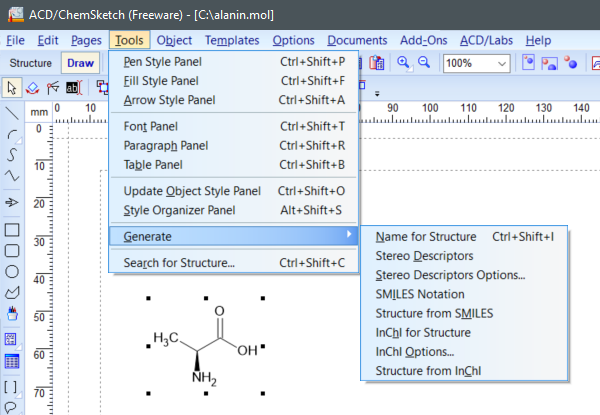
(ACD/ChemSketch v2021.1.1, Advanced Chemistry Development, Inc., Toronto, ON, Kanada, 2021).
Wenn Sie InChI für Structure auswählen, wird auch der InChIKey angezeigt, wenn er unter InChI Options aktiviert ist. Um den InChIKey einzubeziehen, wählen Sie Tools -> Generate -> InChI Option und markieren Sie InChI key.
MarvinSketch
Um SMILES, InChI und InChIKey in MarvinSketch abzurufen, markieren Sie eine Struktur und wählen dann Edit -> Copy As. In einem neuen Fenster wählen Sie Daylight SMILES, InChI/RInChI oder InChIKey/RInChIKey.
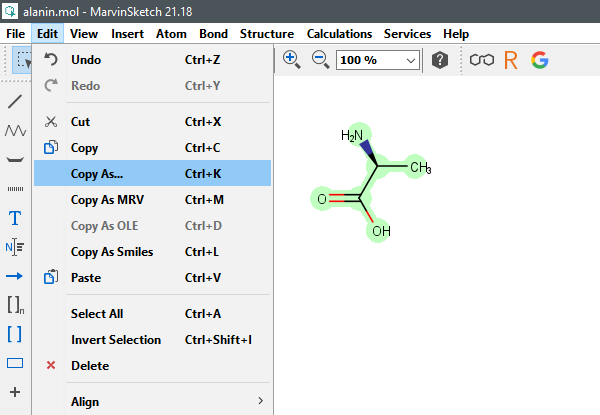
(MarvinSketch v21.18, ChemAxon, Ltd., Budapest, Ungarn, 2021.)
Alternativ können SMILES, InChI und InChIKey auch als Textdateien gespeichert werden, indem Sie File -> Save as -> Daylight SMILES, InChI/RInChI oder InChIKey/RInChIKey aus dem Dropdown-Menü wählen und -> Speichern.
Was brauchen Sie und wann?
CT-Dateien, SMILES und InChI sind unterschiedliche Repräsentation von chemischen Strukturen. Ein wichtiges Merkmal von CT-Dateien ist die Möglichkeit, 3D-Daten von Molekülen zu speichern. CT-Dateien sind die richtige Wahl, wenn es darum geht, die 3D-Struktur von Molekülen in maschinenlesbarer Form zu beschreiben, die z. B. mit Hilfe der Einkristall-Röntgenbeugung (XRD) gewonnen wurde. Für alle anderen Anwendungsfälle sind InChI und SMILES ausreichend, mit dem zusätzlichen Vorteil, dass es sich um (einfache) Zeilennotationen handelt.
Bitte beachten Sie auch, dass InChI ein Identifikator ist, während SMILES ein Strukturcode ist. Eine Umwandlung eines SMILES-Codes in eine graphische Darstellung einer chemischen Struktur und zurück ist mit SMILES möglich, wobei dies nicht notwendigerweise denselben SMILES-Code liefert, wie den ursprünglich bereitgestellten, weshalb SMILES kein Identifikator ist. Andererseits ist InChI ein Identifikator und nicht dazu gedacht, die korrekte chemische Strukturzeichnung zu regenerieren, da InChI Konnektivitäten kennt, nicht aber die Bindungsordnungen.
SMILES und InChI sowie InChI Key sind ideal, um chemische Strukturen aus Forschungsartikeln in einer ergänzenden Tabelle in maschinenlesbarer Art zu beschreiben. Eine solche Tabelle sollte Teil eines Datensatzes in einem generischen Repositorium sein, könnte aber auch zusammen mit dem Manuskript beim akademischen Verlag eingereicht werden, wie es beim ACS Journal of Medicinal Chemistry seit 2014 für SMILES verlangt wird.
Maschinenlesbare Daten als ergänzende Tabelle bereitstellen
Informationen zur Verbesserung der maschinellen Lesbarkeit könnten in einem Datensatz als ergänzende Tabelle bereitgestellt werden, da es noch nicht für alle Arten von Chemiedaten offene Formate gibt, die diese Informationen enthalten. Diese Tabelle sollte auch in einem maschinenlesbaren, textbasierten Format wie z. B. einer CSV-Datei bereitgestellt werden.
Die minimalen und empfohlenen Spalten einer solchen Tabelle in der Chemie lauten wie folgt:
- Buchstabencode und Nummer in Ihrem Laborjournal, d.h. ein lokaler Probenidentifikator
- Nummer(n) der Strukturen im Artikel, d. h. ein lokaler Strukturbezeichner innerhalb Ihres (korrespondierenden) Artikels
- SMILES
- InChI
- InChIKey
Der Buchstabencode und die Nummer in Ihrem Laborjournal sollten enthalten sein, da analytische Daten häufig nach den Einträgen im Laborjournal benannt werden.
Zusätzlich kann diese Tabelle noch weitere Spalten enthalten für:
- CAS-Registernummer
- IUPAC-Name
- Trivialnamen einer Verbindung
- PubChem-Verbindungsidentifikator (PubChem CID)
- CAS-Registernummer
- Kommentar (falls erforderlich)
Vorlagen für eine solche Tabelle werden als ODS, XLSX und CSV bereitgestellt. Diese Vorlagendateien nutzen auch die Vorteile von Ontologien, um Begriffe für Menschen und Maschinen eindeutig zu identifizieren.
Wenn die experimentelle Arbeit in einem ELN dokumentiert ist, kann eine solche Tabelle auch vom ELN-System bereitgestellt werden. Chemotion ELN generiert SMILES, InChI, InChIKey sowie RInChI und RInChIKey für Verbindungen und Reaktionen. Diese Informationen sind auch im Chemotion Repository verfügbar, d.h. eine solche Zusatztabelle ist bei strukturierten domänenspezifischen Repositorien, wie Chemotion Repository, nicht erforderlich, während Datensätze in generischen Repositorien von einer solchen Tabelle profitieren.
Diese Seite ist lizenziert unter einer Creative Commons Universal (CC0 1.0) Public Domain Dedication International Lizenz._
Hauptbeitragende: ORCID:0000-0003-4480-8661