Best Practice
Auf dieser Seite stellen wir bewährte Verfahren für die Veröffentlichung von Daten in fachspezifischen und generischen Repositorien für Forschungsdaten vor.
Best-Practice-Beispiele für fachspezifische Repositorien
Für die fachspezifischen Repositorien Chemotion Repository in Kombination mit Chemotion ELN werden die Daten entlang der wissenschaftlichen Arbeitsabläufe gesammelt, Analytikdaten automatisch in offene Formate konvertiert, kontrollierte Vokabulare oder Ontologien zur Datenbeschreibung verwendet und Analytikdaten mit Reaktionen und Proben, also chemischen Strukturen, verknüpft. Das gesamte Chemotion-Paket ermöglicht es Forschenden, verschiedene Arten von Analytikdaten, die mit Reaktionen und Proben verbunden sind, in einer [digitalen Umgebung] zu sammeln, zu verarbeiten, zu analysieren, zu speichern und zu veröffentlichen (/docs/smartlab). Die FAIRifizierung von Daten entlang dieses Datenlebenszyklus wird hauptsächlich von Chemotion übernommen. Beim nahtlosen Export aus dem ELN in das Repository werden den hinterlegten Daten PIDs (in diesem Fall DOIs) zugewiesen. Chemotion Repository vergibt DOIs für Reaktionen, Proben, Analysedaten und Sammlungen (Sammel-PIDs).
Andere feldspezifische Repositorien wie CSD und ICSD, Strenda DB, SupraBank, NOMAD und Chemotion Repository, bieten, wenn sie unabhängig von Chemotion ELN verwendet werden, integrierte Arbeitsabläufe für die Aggregation strukturierter Daten entlang der Einreichungswege, die mit den FAIR Data Principles übereinstimmen. Dadurch wird die Veröffentlichung von FAIRen-Daten erheblich erleichtert.
Weitere zu berücksichtigende Aspekte sind die Bereitstellung umfangreicher Metadaten, bei denen es sich um fachunabhängige Metadaten oder fachspezifische Metadaten handeln kann, und die auch Informationen zur Provenienz enthalten sollten, die bisher typischerweise im ergänzenden PDF-Abschnitt zu allgemeinen Informationen und Protokollen enthalten waren. Die Mindestinformationsanforderungen sollten befolgt werden, wie sie von der Chemiegemeinschaft festgelegt und in Artikeln und früher auch in ergänzenden PDFs umgesetzt wurden.
Fragen Sie sich jetzt gerade: "Das war's?" Ja, das ist es! Wie bereits erwähnt, übernehmen fachspezifische Repositorien einen Großteil der schweren Arbeit, wenn es um die FAIRifizierung geht, während eine digitale Smart-Lab-Umgebung sogar strukturierte Daten direkt entlang der wissenschaftlichen Arbeitsabläufe und des Datenlebenszyklus sammelt.
Best-Practice-Beispiele für fachspezifische Repositorien
Zum besseren Verständnis der Datenveröffentlichungen in fachspezifische Repositorien und der Verknüpfung dieser Daten mit den entsprechenden Zeitschriftenveröffentlichungen können die folgenden Beispiele einschließlich ihrer Daten über die folgenden Seiten "Lead by Example" aufgerufen werden:
- im Chemotion Repository: Modulare Synthese von neuen Pyrrolochinolinchinon-Derivaten.
- in Chemotion Repository und CSD: Modulare Synthese von trans-A2B2-Porphyrinen mit terminalen Estern: Systematische Erweiterung des Spektrums an linearen Linkern für Porphyrin-basierte MOFs.
Bewährte Praktiken für generische Repositories
Während fachspezifische Repositorien die FAIRifizierung von Daten entlang des gesamten Datenlebenszyklus erheblich unterstützen, erfolgt die Vorbereitung von Datensätzen für die Datenveröffentlichung in allgemeinen Repositorien in der Phase der Offenlegung und Veröffentlichung, solange keine (institutionellen) FDM-Workflows die Datenveröffentlichung unterstützen. In der Regel werden die Daten gleichzeitig mit der Vorbereitung eines entsprechenden Manuskripts gesammelt. Daher verlangen Datensätze, die über ein allgemeines Repository veröffentlicht werden sollen, eine manuelle FAIRifizierung vor dem Hochladen und der Veröffentlichung.
Zunächst sollten alle zu veröffentlichenden Daten in einer logischen Ordnerstruktur gesammelt und geordnet werden, z. B. ein Ordner für NMR- und ein weiterer für MS-Daten (Abbildung 1). Verschachtelte Ordnerstrukturen sollten vermieden und Aspekte der Datenorganisation, wie z. B. die Dateibenennung, berücksichtigt werden. Forschende sollten ein Datenpaket anstreben, das so selbsterklärend ist wie die ergänzenden PDFs, die sie zuvor zusammen mit wissenschaftlichen Artikeln veröffentlicht haben.
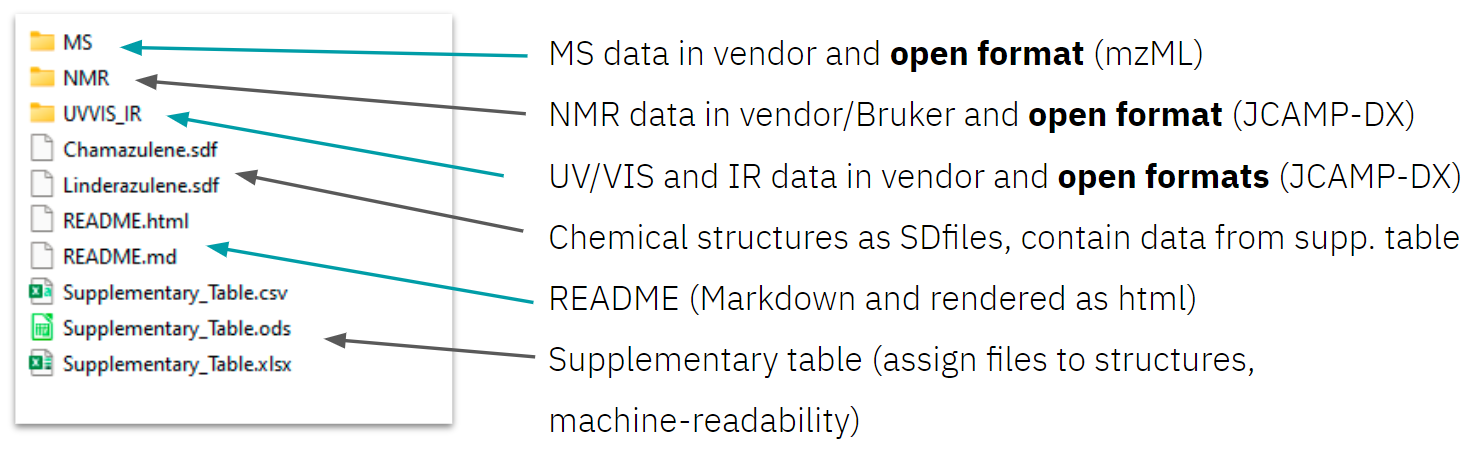
Abbildung 1: Beispiel für die Ordnerstruktur und den Inhalt des Lead by Example Datensatzes von Linderazuelen.
Um weitere Aspekte zu berücksichtigen, stellen wir eine nicht erschöpfende Liste allgemeiner Best Practices zur Verfügung, die zeigen, was ein Chemiedatensatz enthalten sollte und wie die chemischen Daten dargestellt und beschrieben werden sollten. Bitte beachten Sie, dass bei der Datenveröffentlichung auch fachspezifische Aspekte berücksichtigt werden müssen.
- Daten sollten in offenen Formaten zusammen mit den ursprünglichen Rohdaten in proprietären Formaten veröffentlicht werden.
Offene Formate für analytische Daten sollten die erste Wahl sein. Viele Analytikinstrumente liefern jedoch Daten in proprietären Formaten. Je nach Spezifikation des verwendeten Formats sind nicht unbedingt alle Daten in diesen Formaten auch in dem ausgewählten offenen Format enthalten, in das sie konvertiert wurden. Daher sollten auch die ursprünglichen Rohdaten (in proprietären Formaten) veröffentlicht werden, auch wenn diese Daten möglicherweise nur begrenzt interoperabel sind. Die Veröffentlichung von Original-Rohdaten ist eine Maßnahme der wissenschaftlichen Integrität und ermöglicht eine unvoreingenommene Weiterverarbeitung und Wiederverwendung von Daten. Wenn kein offenes Format vorhanden ist, sollte der Export als Textdatei, d. h. ohne jegliche Formatspezifikation, in Betracht gezogen werden. - Daten sollten mit chemischen Strukturen und Reaktionen verknüpft sein.
Da analytische Daten in der Regel nach Laborjournaleinträgen benannt werden, muss der Datensatz eine Beschreibung enthalten, welche Daten welcher Probe, welchem Molekül oder welchem Experiment zugeordnet sind. Dies sollte in dem für die analytischen Daten verwendeten Format angegeben werden. Eine andere Lösung ist die Bereitstellung einer Zusatztabelle (Abbildung 1) innerhalb des Datenpakets, die InChI-Strukturidentifikatoren und SMILES-Strukturcodes sowie zusätzliche Informationen wie RInChI-Reaktionsidentifikatoren enthalten sollte. Chemische Strukturen der zweiten Ebene können ebenfalls hinzugefügt und als CT-Dateien wie mol oder SDfiles dargestellt werden (Abbildung 1). RXNfiles kann verwendet werden, um chemische Reaktionen in maschinenlesbarer Form zu beschreiben. - Datensätze sollten Skripte und Arbeitsabläufe sowie Informationen über die verwendete Software enthalten.
Arbeitsabläufe, d. h. Skripte für die Datenverarbeitung und Eingabeparameter für halbautomatische Skripte, sollten enthalten sein. Die Metadaten des gesamten Datensatzes sollten die Sprache und die Version der verwendeten Skripte sowie weitere verwendete Software beschreiben. Am besten ist es, nicht nur den Code einzubinden, sondern auch Notebooks wie Form von R Notebooks oder Jupyther Notebooks hinzuzufügen. - Daten sollten mit Metadaten beschrieben werden, und es sollten Mindestinformationsstandards eingehalten werden.
Fachspezifische Metadaten sollten über den Metadaten-Editor des Repositoriums bereitgestellt werden. Zusätzlich sollten fachspezifische Metadaten über den Metadaten-Editor veröffentlicht werden, sofern dies vom gewählten Repositorium unterstützt wird, alternativ in den analytischen Daten oder einer zusätzlichen README-Datei. Die Mindestinformationsanforderungen, wie sie von der Chemiegemeinschaft festgelegt wurden und in Manuskripten und früher auch in ergänzenden PDFs befolgt wurden, sollten enthalten sein. - Die Informationen zur Provenienz der Daten sollten enthalten sein.
Ein Teil dieser Herkunftsinformationen ist Teil der Metadaten des Datensatzes und sollte über den Metadaten-Editor des Repositorys hinzugefügt werden. Alle Informationen, die bisher typischerweise in den ergänzenden PDFs im Abschnitt über allgemeine Informationen und Protokolle aufgenommen wurden, z. B. Informationen über verwendete Methoden und Instrumente, sind Herkunftsinformationen und sollten in eine README-Datei aufgenommen werden, sofern kein domänenspezifisches Metadatenschema verfügbar ist. Diese README könnte eine Textdatei sein oder in Markdown geschrieben werden, wobei auch eine menschenlesbare Darstellung als HTML bereitgestellt werden könnte.
Klingt das für Sie nach viel manueller Arbeit? Vermeiden Sie zusätzliche Arbeit, indem Sie eine digitale Smart Lab Umgebung zum Sammeln, Verarbeiten, Analysieren und Veröffentlichen von Forschungsdaten nutzen, wie z. B. Chemotion ELN in Kombination mit Chemotion Repository! Außerdem können Sie auf die der Erstellung Zusatzinformationen als PDFs verzichten und die eingesparte Arbeitszeit nutzen, um Ihren Datensatz für die Veröffentlichung vorzubereiten!
Best-Practice-Beispiele für generische Repositorien
Zum besseren Verständnis der Datenveröffentlichungen in generischen Repositorien und der Verknüpfung dieser Daten mit entsprechenden Zeitschriftenveröffentlichungen können die folgenden Beispiele einschließlich der zugehörigen Daten über die folgenden Lead-by-Example-Seiten aufgerufen werden:
- in RADAR4Chem: Linderazulen aus einer invasiven Pflanze - Delphi und sein violettes Wunder.
- in Zenodo: A dataset of 255,000 randomly selected and manually classified extracted ion chromatograms for evaluation of peak detection methods.
- in RADAR: Synthesis and biological evaluation of highly potent fungicidal deoxy-hygrophorones.
- in DaRus: Predictive design of ordered mesoporous silica with well-defined, ultra-large mesopores.
Hauptbeitragende: ORCID:0000-0003-4480-8661, ORCID:0000-0002-6243-2840 and ORCID:0000-0003-2060-842X